
Typage ADN et ses applications dans les contrôles de filiation
Outil d'une grande fiabilité, le typage ADN est devenu un élément essentiel de l'identification des équidés. Il permet notamment de procéder au contrôle de filiation, afin de s'assurer de la génétique d'un individu.
- Les microsatellites : supports du typage ADN
- Analyse du génotype
- Utilisation des génotypes pour contrôler les filiations
- Dans la pratique
Les microsatellites : supports du typage ADN
Chaque être vivant a le privilège d’être unique, distinct de ses congénères. Il est l’expression de l’information codée par son génome : la longue molécule d’ADN contenue dans le noyau de chacune de ses cellules. C’est sur ce principe que repose l’identification d’un individu et son application directe, le contrôle de filiation.
L'ADN est constitué de plusieurs composants chimiques notamment de bases azotées. Il existe 4 bases différentes (adénine A, cytosine C, guanine G et thymine T), dont l'assemblage avec les autres constituants forme les nucléotides (schéma 1).

Sur la longue molécule d'ADN, support de l'hérédité, on distingue des régions qui codent pour des protéines (les gènes) et d’autres régions non codantes. Dans ces dernières, il existe des régions appelées microsatellites qui sont constituées de répétitions de séquences de 2, 3 ou 4 nucléotides. On estime entre 50 000 à 100 000 le nombre de microsatellites dans le génome. Les plus fréquemment utilisés chez les équidés sont des répétitions de 2 nucléotides.
Pour un microsatellite donné :
- il existe différents allèles définis par le nombre de ces répétitions, variable d'un individu à l'autre (ex. : 6 répétitions définiront l’allèle A, 8 répétitions définiront l’allèle B, …) (Schéma 2) ;
- ce nombre est stable au cours de la vie de l’animal et se transmet à sa descendance.
Chez chaque individu, un microsatellite possède deux allèles (variants) : l’un d’origine paternelle, l’autre d’origine maternelle. Lorsque les deux allèles sont identiques, l’individu est qualifié d’homozygote ; lorsqu’ils sont différents, il est hétérozygote.
L’ensemble des allèles d’un microsatellite constitue le polymorphisme génétique. Plus le nombre d’allèles sera élevé, plus le microsatellite sera efficace pour identifier un animal et le distinguer des autres. Le typage de l’ensemble des microsatellites étudiés définit le génotype de l’individu.
Analyse du génotype
Les méthodes d’étude des microsatellites sont reproductibles, automatisables et très précises, donc fiables et efficaces. Les techniques de biologie moléculaire mises en oeuvre permettent d’effectuer les étapes indispensables à l'analyse.
1ère étape : extraction de l’ADN
Toute cellule possédant un noyau contient de l’ADN nucléaire. C’est notamment le cas des globules blancs, seules cellules sanguines des équidés possédant un noyau. On peut également extraire l’ADN de nombreuses autres cellules (bulbes pileux, sperme, cellules buccales…).
La prise de sang est la technique la plus utilisée pour obtenir de l'ADN car elle offre un bon compromis entre :
- facilité du prélèvement,
- fiabilité de l'échantillon,
- automatisation des résultats.
Cet ADN peut être conservé plusieurs années et être réutilisé pour d’autres études.
2ème étape : amplification des microsatellites de l’ADN
La polymerase chain reaction (PCR) ou "réaction de polymérisation en chaîne" est une technique de biologie moléculaire assurant l'amplification in vitro d'un fragment d'ADN jusqu'à un milliard de fois.
Cette technique peut s’apparenter à une "photocopie" en un très grand nombre d’exemplaires d’un fragment d’ADN contenant les microsatellites à analyser.
La taille de l'amplifiat varie en fonction du nombre de répétitions de séquences de nucléotides. A chaque taille possible correspond un allèle du microsatellite.
3ème étape : séparation des différents allèles par électrophorèse
Ces amplifiats sont séparés en fonction de leur taille. Toutes les données sont intégrées par un ordinateur muni d'un logiciel d’analyse.
Etape finale : Résultat de l’analyse du génotype
Les premiers microsatellites équins ont été mis en évidence en 1992 par une équipe suédoise et ont ouvert la voie à de nombreuses recherches menées par différentes équipes internationales. Ces dernières ont également mis en évidence que ces microsatellites équins étaient représentés dans le génome des espèces du genre Equus (ânes, zèbres, Przewalski).
Cette collaboration menée sous les auspices d’une société savante internationale, l’ISAG (international society for animal genetics), a permis d’établir un panel international de typage composé de 12 microsatellites. Ce panel est utilisé aujourd’hui par plus de 60 laboratoires à travers le monde. Il est contrôlé tous les deux ans par un test de comparaison international et permet à de nouveaux laboratoires de rejoindre ce groupe.
Les laboratoires d’analyse ont toute liberté d’ajouter des microsatellites à ce panel. Ainsi, l’identification d’un cheval en France est réalisée, depuis l’année 2000, à l’aide de 14 microsatellites.
| Panel international de 12 microsatellites | AHT 4 VHL 20 AHT5 HTG 4 ASB 2 HTG 10 ASB 17 HMS 3 ASB 23 HMS 6 HMS 2 HMS 7 |
|---|---|
| 2 microsatellites supplémentaires utilisés en France | HTG 6 HMS 1 |
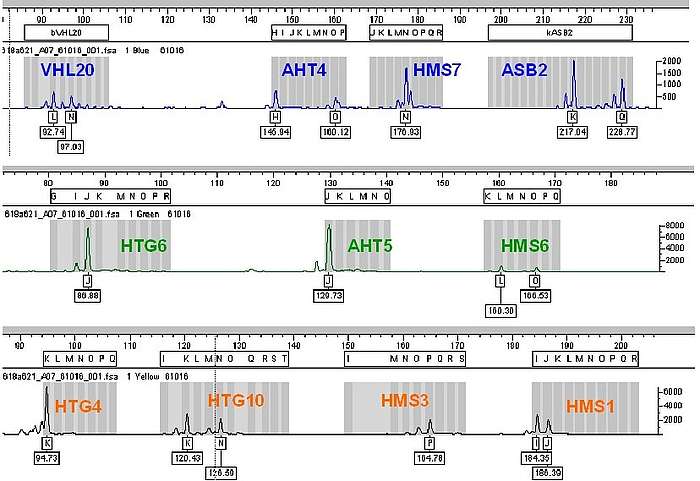
Chaque allèle, initialement interprété en taille, est ensuite transformé en lettre selon une nomenclature internationale. Lors de l'identification par typage ADN d'un équidé, un génotype se présente donc sous la forme décrite au schéma 3 :
- Dans cet exemple on notera que, pour le 1er microsatellite noté VHL20, le cheval analysé est hétérozygote L N, puisqu’il a reçu l’allèle L d’un de ses parents et l’allèle N de son autre parent.
- Pour le microsatellite noté HMS7, l’animal est homozygote puisqu’il a reçu le même allèle N de son père et de sa mère.
Utilisation des génotypes pour contrôler les filiations
L'utilisation des génotypes est définie réglementairement par l’arrêté du 24 avril 2009, relatif à l’identification et aux contrôles de filiation des équidés par typage ADN. Il est éventuellement précisé par les règlements de stud-books.
Principe
Après établissement des génotypes, le contrôle de filiation consiste à comparer le génotype d’un produit avec celui de ses parents présumés.
Pour chaque microsatellite, le produit doit avoir hérité d'un allèle d’origine paternelle et d'un autre d’origine maternelle. Si tel n’est pas le cas, la filiation est déclarée incompatible.
A l’inverse, si aucune incompatibilité n’est détectée sur l’ensemble des 14 microsatellites, la filiation est déclarée compatible.
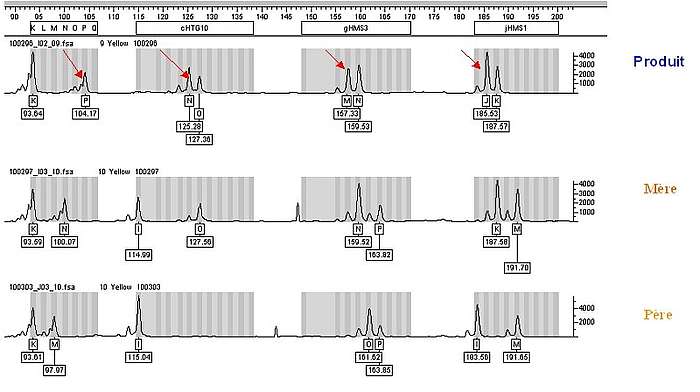
Le contrôle de filiation, présenté dans l'exemple du schéma 4, indique une incompatibilité avec le père sur 4 microsatellites : les allèles P (HTG4), N (HTG10), M (HMS3) et J (HMS1) présents chez le produit ne peuvent avoir été transmis par le père et permettent de déclarer la filiation incompatible de père.
Garantie de l’identité de l’individu
Pour un microsatellite, il existe un nombre d'allèles (variants) différent pour chaque race, ainsi qu'une fréquence d'apparition pour chaque allèle. Ceci permet de chiffrer la probabilité d’avoir deux animaux identiques.
Il s’agit de calculer la probabilité de tirer deux individus au hasard ayant le même génotype dans une population donnée (race). On peut rapidement se rendre compte que la probabilité de tirer deux individus identiques dans les populations citées est quasi nulle (tableau 2). Chez le Pur sang, par exemple, une probabilité de 4,6.10-10 signifie qu’il n’y a pas deux individus identiques sur 250 millions de chevaux, ce qui excède très largement la taille de la population mondiale.
| Pur Sang | Arabe | |
|---|---|---|
| Microsatellites | Nombre d'allèles | Nombre d'allèles |
| AHT4 | 6 | 7 |
| AHT5 | 6 | 6 |
| ASB2 | 8 | 8 |
| HMS1 | 4 | 6 |
| HMS3 | 6 | 6 |
| HMS6 | 7 | 6 |
| HMS7 | 5 | 7 |
| HTG4 | 5 | 6 |
| HTG6 | 7 | 7 |
| HTG10 | 7 | 8 |
| VHL20 | 7 | 10 |
| Total | 68 | 77 |
| Probabilité d'identité | 4,6 10-10 | 1,8 10-12 |
Tableau 2 : Probabilités d'exclusion et d'identité selon les microsatellites dans 2 races de chevaux de sang.
Dans la pratique
Un prélèvement de sang (un tube de 5 ml contenant une solution anticoagulante d’EDTA de potassium) est nécessaire et suffisant pour chaque équidé à analyser. Il doit être effectué par une personne habilitée à l'identification (agent Ifce, vétérinaire agréé…).
Le matériel du laboratoire doit impérativement être demandé avant toute intervention.
Chaque échantillon doit être correctement identifié, accompagné du formulaire de signalement ou d’un bordereau du laboratoire mentionnant l’état-civil de l’animal prélevé ainsi que son numéro SIRE, et adressé au laboratoire.
Outils remarquablement fiables et efficaces, l’identification génétique par les microsatellites et le contrôle de filiation permettent de rendre des services quotidiens aux acteurs de la filière équine. De plus, ces méthodes internationales autorisent des échanges de données permettant un meilleur suivi des équidés à travers le monde.

En savoir plus sur nos auteurs
- Clothilde DUBOIS Formatrice IFCE
- JC. MERIAUX Labogena
- Sophie DANVY Ingénieur de développement IFCE
- Margot SABBAGH Ingénieure de projets & développement « Génétique des équidés » IFCE