
L'utilisation des puces à ADN
Les puces à ADN permettent des tests rapides, sensibles et spécifiques. Leur utilisation se développe : petit tour d'horizon de quelques possibilités qu'elles offrent.

- Mode de fonctionnement des puces à ADN
- Utilisation pour la détection d’agents pathogènes
- Utilisation pour la mesure de l’expression des gènes (transcriptomique)
- Utilisation pour l'étude du génome (génomique)
- En conclusion
Mode de fonctionnement des puces à ADN
Le concept des puces à ADN est né dans les années 1990. Il repose sur la combinaison de plusieurs disciplines telles que la micro-électronique, la chimie des acides nucléiques, l’analyse d’image et la bioinformatique.
Le principe de la puce à ADN est basé sur la complémentarité des deux brins de l’ADN et donc de façon plus générale sur la technique d’hybridation. Ces puces permettent des tests plus rapides, plus sensibles et plus spécifiques. En évitant certaines étapes préliminaires telle que la culture, cela permet d’obtenir un résultat en quelques heures là où plusieurs jours étaient auparavant nécessaires.
Schématiquement, les puces à ADN sont des supports (lame de verre ou de silicium, de différente taille) sur lesquels sont régulièrement répartis des morceaux d’ADN simple brin, aussi appelés sondes et dont l’enchaînement des bases est connu. La cible est l’ADN simple brin qui doit être analysé, car l’enchaînement de ses bases est inconnu. Avant d’être déposé sur la puce, il est marqué à l’aide d’une molécule fluorescente.
Lorsque la cible est mise en contact avec la sonde, seuls les bouts d’ADN complémentaires vont s’hybrider. La puce est ensuite lavée plusieurs fois afin qu’il ne reste sur la lame que les brins qui se seront parfaitement appariés. A l’endroit où les deux brins d’ADN s’apparient, il apparaît un spot lumineux.
Actuellement, il existe 3 types de puces qui diffèrent par le nombre de sondes qu’elles renferment :
- Les puces à basse densité
- Les puces à densité moyenne, utilisées pour mesurer l’expression de gènes
- Les puces à haute densité permettent l’analyse de génomes complets
Utilisation pour la détection d’agents pathogènes
Exemple de la puce à ADN développée au laboratoire Frank Duncombe
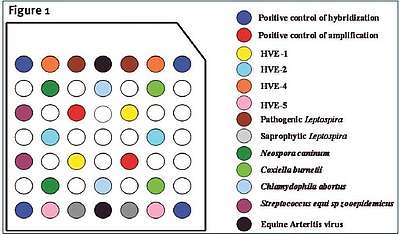
On obtient ainsi un profil différent en fonction des pathogènes présents ou non dans les échantillons analysés (Figure 1).
Le choix des pathogènes, pour cette première version de puce à ADN, résulte d’une réflexion qui nous a conduit à prendre en considération d’une part les données de la littérature (Herpès virus équin de type 1 et Streptoccocus equi subsp zooepidemicus : 2 agents abortifs majeurs), et d’autre part une étude expérimentale sur de nouveaux pathogènes que nous avons conduite précédemment. Ainsi, les pathogènes retenus sont : HVE-1, HVE-2, HVE-4, HVE-5, leptospires pathogènes, Coxiella burnetii, Chlamydophila abortus, Streptoccocus equi subsp zooepidemicus, le virus de l’Artérite virale équine et Neospora caninum. A chaque pathogène correspondent deux spots spécifiques sur le support de la puce.
En amont de cette révélation sur puce, deux systèmes d’amplification simultanée de plusieurs pathogènes (PCR multiplexe) ont été optimisés sans perte de sensibilité ni de spécificité par rapport aux tests individuels. La spécificité de ces systèmes a également été testée par des tests d’inclusivité et exclusivité.
A ce jour, une étude comparative a été réalisée entre la détection par puce à ADN et les analyses réalisées en diagnostic de première intention (HVE-1, HVE-4, AVE et leptospire), sur 216 avortons. Le tableau ci-dessous résume les résultats obtenus.
Récapitulatif des résultats de la première étude comparative entre PCR réalisées individuellement et révélation sur puce ADN
| Diagnostic de 1ère intention | Total | |||
| Positif | Négatif | |||
| Puce à ADN | Positif (%) | 35 (19,3%) | 24 (13,2%) | 59 |
| Négatif (%) | 1 (0,5%) | 122 (67%) | 123 | |
| Total | 36 | 146 | 182 | |
Pour 157 cas sur 182, les résultats sont concordants entre les deux types d’analyses. Pour 25 cas sur 182 des différences ont été observées :
- Un cas positif en diagnostic de 1ère intention (PCR) était négatif suite à l’analyse sur puce. L’analyse sur puce ayant été réalisée après conservation à -80°C, une possible dégradation des acides nucléiques est suspectée.
- 24 cas négatifs en analyse de 1ère intention étaient positifs suite à l’analyse sur puce. L’essentiel de ces divergences s’explique par l’évolution des techniques PCR employées.
Ces résultats se traduisent par une sensibilité de 97% et une spécificité de 83% pour la puce à ADN.
Pour 34 cas, la puce a permis de mettre en évidence un pathogène supplémentaire non recherché en diagnostic de première intention mais dont la présence a pu être ensuite confirmée par une PCR individuelle spécifique. La révélation sur puce a permis aussi de mettre en évidence des co-détections.
Les biopuces permettent la détection simultanée de plusieurs pathogènes. Toutefois, leur miniaturisation pourrait permettre une automatisation. A ce jour, l’amplification est le préalable obligatoire à toutes détections sur puce. En effet, le seuil de sensibilité de la technique ne permet pas encore de s’en affranchir. C’est cependant dans cette direction que convergent aujourd’hui les travaux de recherches dans ce domaine.
De plus, le caractère totalement évolutif de ce système permet d’envisager demain sans difficulté l’incorporation d’autres pathogènes à notre puce à ADN dédiée aux avortements équins, de même que la déclinaison de ce nouvel outil à d’autres pathologies.
Utilisation pour la mesure de l’expression des gènes (transcriptomique)
Certaines puces permettent de détecter les gènes surexprimés ou sous-exprimés dans certaines conditions (maladie, traitement…), grâce à la détection des ARN messagers extraits des cellules.
Utilisation pour l'étude du génome (génomique)
Le séquençage complet du génome équin a été réalisé en 2006 aux Etats-Unis par le Broad Institute et annoncé officiellement le 7 février 2007. Il mesure environ 2,7 gigabases (Gb), une taille légèrement inférieure à celle de l’Homme (2,9 Gb). L’analyse plus fine de ce génome prédit l’existence d’un peu plus de 20 000 gènes codant pour des protéines dont environ 17 000 sont similaires de gènes de l’Homme, de la souris et du chien.
Une puce à ADN permettant un génotypage rapide et précis a été mise au point, et est désormais commercialisée. Le substrat de départ n’est plus l’ARN mais l’ADN du cheval. Contenant plus de 54 000 marqueurs génétiques de type SNP (Single Nucleotide Polymorphism), cette puce permet d’envisager des études génétiques complexes jusque là inaccessibles à la recherche équine.
En conclusion
L’utilisation de la nanotechnologie dans le diagnostic vétérinaire n’en est qu’à ses débuts. Avec les puces à basse densité dédiées aux avortements, le délai de réponse rapide donne au vétérinaire la possibilité de prendre des mesures sanitaires pour prévenir toute contamination transversale ou épidémie au sein d’un effectif de juments.
Les puces à moyenne et haute densités produisent d’énormes quantités de données qui croissent exponentiellement avec le temps. Les résultats rapidement obtenus et en quantité importante nécessitent des outils informatiques puissants pour les traiter, les organiser et les analyser.
La rapidité de l’évolution dans ce domaine suggère que de nouvelles applications en santé équine seraient susceptibles d’apparaître dans les années à venir (séquençage par hybridation, la pharmacogénomique, la toxicogénomique…).

En savoir plus sur nos auteurs
- Albertine LÉON Chef de service - Labéo Frank Duncombe
- B. BLANCHARD
- T. ROUILLARD
- S. PRONOST R&D Labéo Frank Duncombe
- G. FORTIER